Des chercheurs de l’Inserm et de l’Université Pierre et Marie Curie du Centre de Recherche des Cordeliers, en collaboration avec des chercheurs de l’institut Karolinska en Suède, ont décrit le mécanisme par lequel le facteur GPS2 réprime la survenue du diabète de type 2 chez les personnes obèses. Ces travaux, publiés dans la revue Nature Medicine, mettent en lumière l’importance de l’épigénome dans l’apparition de la maladie.

Fréquemment associés, l’obésité et le diabète de type 2 sont des pathologies sévères qui sont en constante progression dans le monde et particulièrement en France. Chez les personnes obèses, l’excès de tissu adipeux s’accompagne d’une inflammation locale, qui participe au développement du diabète de type 2 et d’autres complications. Ce phénomène résulte d’une activation anormale des cellules immunitaires, notamment les macrophages qui produisent des facteurs propices à l’inflammation.
Un des mécanismes qui contrôle l’expression de certains gènes, dont ceux impliqués dans l’inflammation, est la modification de la structure de la chromatine (épigénome). En effet, celle-ci doit être décompactée afin d’être accessible à des complexes protéiques qui permettent aux gènes de s’exprimer. Les équipes de Nicolas Venteclef et de Eckardt Treuter ont décrit le rôle important du régulateur transcriptionel GPS2 dans le contrôle de l’activation des macrophages par la modification de l’épigénome, au cours de l’obésité et du diabète de type 2.
En comparant les tissus adipeux de personnes obèses et non-obèses, les chercheurs ont montré que le statut diabétique coïncide avec une faible expression de GPS2 dans les macrophages, associée à une inflammation élevée. Afin de confirmer la relation de cause à effet, ils ont généré des souris déficientes en GPS2 spécifiquement dans les macrophages, et les ont mises sous régime riche en graisses pour provoquer l’obésité et le diabète de type 2. Comme observé chez l’homme, la déficience en GPS2 amplifie la réponse inflammatoire et le profil diabétique (résistance à l’insuline) liés à l’obésité. A l’inverse, lorsque l’expression de GPS2 est augmentée chez les souris obèses et diabétiques, la progression de la maladie est ralentie. GPS2 réprime donc l’inflammation diabétogène.
Le décodage des mécanismes mis en jeu révèle que GPS2 agit en maintenant la chromatine dans un état transcriptionnel inactif (par le biais de l’acétylation des histones), ce qui empêche l’expression des gènes codant pour des molécules pro-inflammatoires. En modulant la structure de la chromatine, GPS2 contrôle l’activation des macrophages par des régulations dites « épi-génétiques ».
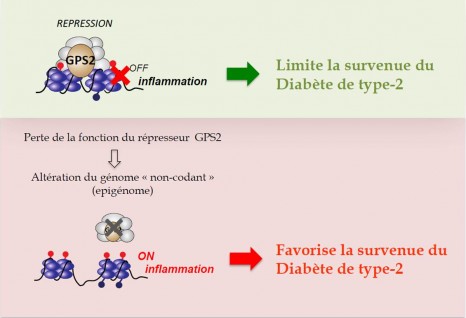
L’ensemble de ce travail montre comment l’épigénome influence la survenue du diabète de type 2 en reprogrammant l’expression des gènes. Cette étude révèle un nouvel aspect important dans la compréhension du diabète lié à l’obésité et ouvre de nouvelles perspectives thérapeutiques pour lutter contre la maladie.
Afin de valider l’influence de l’épigénome sur la survenue du diabète de type-2, une étude sur un plus grand nombre de personnes pré-diabétiques et diabétiques est actuellement en cours au Centre Universitaire du Diabète et de ses Complications (Hôpital Lariboisière, Paris). Cette étude est co-dirigée par le Professeur Jean-François Gauthier et Nicolas Venteclef.