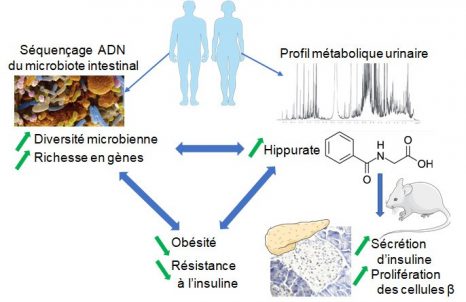
Human and preclinical studies of the host–gut microbiome co-metabolite hippurate as a marker and mediator of metabolic health
François Brial,1 Julien Chilloux,2 Trine Nielsen,3 Sara Vieira-Silva,4 Gwen Falony,4 Petros Andrikopoulos,2,5 Michael Olanipekun,2,5 Lesley Hoyles,6 Fatima Djouadi,7,8 Ana L Neves,2 Andrea Rodriguez-Martinez,2 Ghiwa Ishac Mouawad,1 Nicolas Pons,9 Sofia Forslund,10 Emmanuelle Le-chatelier,9 Aurélie Le Lay,1 Jeremy Nicholson,2 Torben Hansen,3 Tuulia Hyötyläinen,11 Karine Clément,12,13 Matej Oresic,14 Peer Bork,15 Stanislav Dusko Ehrlich,9,16 Jeroen Raes,4,17 Oluf Borbye Pedersen,3 Dominique Gauguier,1,18 Marc-Emmanuel Dumas2,5,18,19
1 UMRS 1124 INSERM, Université de Paris, Paris, France
2 Section of Biomolecular Medicine, Department of Metabolism, Digestion and
Reproduction, Imperial College London, London, UK
3 Novo Nordisk Foundation Centre for Basic Metabolic Research, University of
Copenhagen, Kobenhavn, Denmark
4 Laboratory of Molecular Bacteriology, Department of Microbiology and Immunology,
Rega Institute for Medical Research, Katholieke Universiteit Leuven, Leuven, Belgium
5 National Heart & Lung Institute, Section of Genomic & Environmental Medicine,
Imperial College London, London, UK
6 Department of Biosciences, Nottingham Trent University, Nottingham, UK
7 Centre de Recherche des Cordeliers, Université de Paris, Paris, France
8 Centre de Recherche des Cordeliers, INSERM, Sorbonne Université, Paris, France
9 Metagenopolis, INRAE, Paris, Île-de-France, France
10Forslund Lab, Max Delbrück Centrum für Molekulare Medizin Experimental and
Clinical Research Center, Berlin, Berlin, Germany
11Department of Chemistry, Örebro University, Örebro, Sweden
12INSERM, U1166, team 6 Nutriomique, Université Pierre et Marie Curie-Paris
6, Paris, France
13 Institute of Cardiometabolism and Nutrition (ICAN), Assistance Publique-Hôpitaux
de Paris, Pitié-Salpêtrière Hospital, Paris, France
14 School of Medical Sciences, Örebro Universitet, Orebro, Sweden
15 Structural and Computational Biology Unit, European Molecular Biology
Laboratory, Heidelberg, Germany
16 Center for Host Microbiome Interactions, King’s College London Dental Institute,
London, UK
17 Center for Microbiology, Vlaams Instituut voor Biotechnologie, Leuven, Belgium
18 McGill Genome Centre & Department of Human Genetics, McGill University,
Montréal, Québec, Canada
19European Genomics Institute for Diabetes,INSERM U1283, CNRS UMR8199,
Institut Pasteur de Lille, Lille University Hospital, Unversity of Lille, Lille, France