 © Fotolia
© Fotolia
Un consortium dirigé par des chercheurs de Helmholtz Munich, de l’Université Ludwig Maximilians (LMU), d’Aix Marseille Université et de l’Inserm, en collaboration avec des partenaires internationaux, met en évidence un mécanisme de communication jusqu’alors inconnu entre les bactéries intestinales et les cellules humaines. Les bactéries présentes dans l’intestin humain peuvent directement délivrer des protéines dans les cellules humaines, influençant ainsi activement les réponses immunitaires. Ces résultats révèlent une nouvelle manière dont le microbiome intestinal agit sur le corps humain et pourraient contribuer à mieux comprendre comment les modifications des bactéries intestinales participent au développement de maladies inflammatoires, comme la maladie de Crohn.
Bien que le microbiome intestinal humain soit depuis longtemps associé à des troubles immunitaires, métaboliques et inflammatoires, la plupart des preuves sont corrélatives et les mécanismes moléculaires à l’origine de ces liens restent largement inexplorés.
« Notre objectif était de comprendre comment les bactéries intestinales affectent la biologie humaine, et pas seulement si elles sont liées à des maladies », explique la première auteure de l’étude. « En cartographiant systématiquement les interactions directes entre les protéines des cellules bactériennes et humaines, nous pouvons désormais suggérer des mécanismes moléculaires à l’origine de ces associations. »
Systèmes d’injection de protéines bactériennes dans les cellules de l’intestin sain
L’étude montre que de nombreuses bactéries intestinales courantes et inoffensives possèdent des systèmes de sécrétion de type III, des structures microscopiques ressemblant à des seringues qui peuvent injecter des protéines bactériennes directement dans les cellules humaines. Jusqu’à présent, on pensait que ces systèmes n’existaient que dans les bactéries pathogènes telles que Salmonella.
« Cela change fondamentalement notre vision des bactéries commensales », explique le professeur Pascal Falter-Braun, directeur de l’Institut de biologie des réseaux à Helmholtz Munich et auteur co-correspondant de l’étude avec Andreas Zanzoni, maitre de conférences au laboratoire TAGC (Inserm, Aix-Marseille Université). « Cela montre que ces bactéries non pathogènes ne sont pas seulement des résidents passifs, mais qu’elles peuvent manipuler activement les cellules humaines en injectant leurs protéines dans nos cellules. »
Cartographier la communication entre les bactéries et les cellules humaines
Afin de comprendre le rôle de ces protéines bactériennes dans les cellules humaines, les chercheurs ont cartographié plus d’un millier d’interactions entre les protéines effectrices bactériennes et les protéines humaines, créant ainsi un réseau d’interactions à grande échelle. Leurs analyses ont montré que les protéines bactériennes ciblent préférentiellement les voies humaines impliquées dans la régulation immunitaire et le métabolisme. D’autres expériences en laboratoire ont confirmé que ces protéines peuvent moduler des voies de signalisation immunitaire clés, notamment les réponses NF-κB et cytokines. Les cytokines sont des molécules de signalisation qui aident à coordonner le système immunitaire et à prévenir les réactions excessives pouvant conduire à des maladies auto-immunes. Par exemple, l’inhibition de l’activité de la cytokine facteur de nécrose tumorale (TNF) est un traitement largement utilisé pour la maladie de Crohn.
Liens avec les maladies inflammatoires de l’intestin
Les chercheurs ont également découvert que les gènes codant pour ces protéines effectrices bactériennes sont enrichis dans le microbiome intestinal des patients atteints de la maladie de Crohn. Cela suggère que le transfert direct de protéines des bactéries intestinales vers les cellules humaines pourrait contribuer à l’inflammation intestinale chronique, fournissant ainsi une explication mécanistique potentielle aux liens précédemment observés entre le microbiome et la maladie.
Une nouvelle perspective sur les interactions entre le microbiome et l’hôte
En identifiant un lien moléculaire jusqu’alors méconnu entre les bactéries intestinales et le système immunitaire humain, cette étude fait progresser notre compréhension de la façon dont le microbiome affecte les cellules humaines, faisant passer la recherche de la corrélation à la causalité. Elle soulève également des interrogations intrigantes, telles que la possibilité d’une évolution de ces systèmes d’injection principalement à des fins pathogènes, ou celle d’une existence en faveur de la coexistence commensale, qui aurait ensuite été détournée par des agents pathogènes.
Les recherches futures viseront à déterminer comment les interactions individuelles entre les effecteurs bactériens et l’hôte fonctionnent dans des tissus et des contextes pathologiques spécifiques, dans le but de traduire ces connaissances en stratégies plus précises pour la prévention et le traitement des maladies.






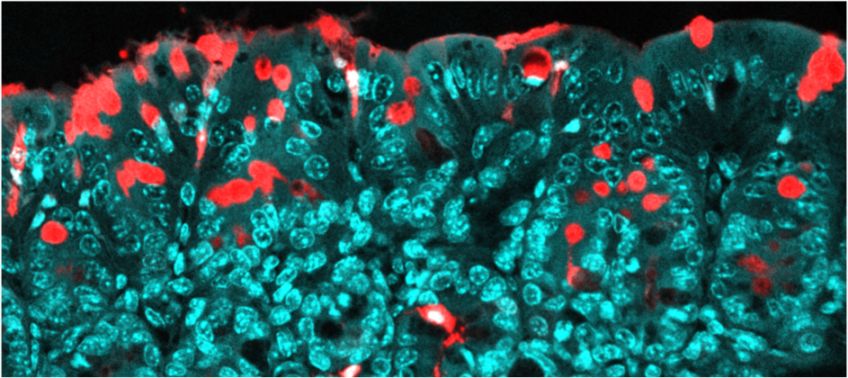 Passage de molécules au travers de l’épithélium du colon, via des voies spécialisées (les cellules de goblet). En rouge : les molécules transportées (antigènes) ; en bleu : l’épithélium. © Institut Pasteur/Interactions Microbiote-Hôte/Clara Delaroque et Benoit Chassaing
Passage de molécules au travers de l’épithélium du colon, via des voies spécialisées (les cellules de goblet). En rouge : les molécules transportées (antigènes) ; en bleu : l’épithélium. © Institut Pasteur/Interactions Microbiote-Hôte/Clara Delaroque et Benoit Chassaing



 L’eczéma du nourrisson se manifeste par des démangeaisons intenses et des rougeurs sur le visage, le cou ou le ventre (image d’illustration). © Adobe stock
L’eczéma du nourrisson se manifeste par des démangeaisons intenses et des rougeurs sur le visage, le cou ou le ventre (image d’illustration). © Adobe stock