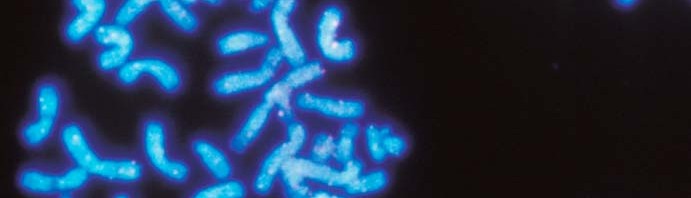
Pourquoi certaines régions chromosomiques sont-elles sensibles à l’apparition de cassures ? Répondre à cette question est crucial, car cette fragilité est impliquée dans le développement de tumeurs. Une équipe de l’Institut de génétique et de biologie moléculaire et cellulaire (CNRS/Inserm/Université de Strasbourg) vient de lever une partie du voile sur ce mystère. Laszlo Tora et ses collègues ont découvert que les cassures au niveau des gènes humains les plus longs sont dues à un phénomène jugé jusqu’ici peu probable dans les cellules des mammifères : une interférence entre deux processus génétiques clefs, la transcription (1) et la réplication de l’ADN (2). Publiés dans le journal Molecular Cell du 23 décembre 2011, ces travaux pourraient mener, à terme, à des stratégies anti-tumorales inédites.
Laszlo Tora et ses collègues ont commencé par étudier la transcription de gènes humains de très grande taille (plus de 800 kilobases -3), connus pour présenter des cassures de l’ADN appelées « sites fragiles communs ». Leur hypothèse de départ : comme le temps requis pour la transcription de ces très grands gènes est extrêmement long, ce processus de transcription pourrait être impliqué dans l’apparition des sites fragiles.
Pour la tester, les chercheurs ont utilisé la technique de cytométrie en flux. Cet outil leur a permis de trier les cellules selon leur avancement dans le cycle cellulaire (4) – cellules en phase G1 (transcription des gènes et croissance de la cellule), S (réplication de l’ADN), G2 (croissance et préparation à la division cellulaire) puis M (division cellulaire). Et, il est apparu que la transcription des très grands gènes dépasse largement la durée du cycle cellulaire, pour se terminer au début du cycle suivant, en phase G1 ou bien S. Un premier résultat étonnant : jusqu’à présent, il était admis, chez les mammifères, que la transcription des gènes avait lieu durant un même cycle cellulaire, et majoritairement en phase G1.
Comme la réplication survient pendant la phase S, les chercheurs ont soupçonné une interférence entre transcription et réplication pour expliquer les cassures sur les très grands gènes des mammifères. Ils ont donc étudié le processus de réplication sur ces gènes. Résultat : la réplication dans la région des sites fragiles survient à la fin de la phase S, alors que la transcription est encore en cours dans ces mêmes régions ! Cette découverte bouscule les connaissances actuelles en génétique. En effet, avant ces travaux, il était généralement admis que les machineries de transcription et de réplication de l’ADN ne pouvaient pas se rencontrer chez les mammifères.
Pour aller plus loin, l’équipe a cherché ensuite à savoir précisément ce qui pouvait fragiliser l’ADN quand réplication et transcription sont concomitantes. Ils ont mis en évidence des structures en boucle qui perdurent, dues à l’hybridation de l’ADN avec la molécule d’ARN produite lors de la transcription. Ce sont ces boucles ADN/ARN qui déstabiliseraient l’ADN jusqu’à provoquer des cassures en cas de stress.
Laszlo Tora et ses collègues ont commencé par étudier la transcription de gènes humains de très grande taille (plus de 800 kilobases -3), connus pour présenter des cassures de l’ADN appelées « sites fragiles communs ». Leur hypothèse de départ : comme le temps requis pour la transcription de ces très grands gènes est extrêmement long, ce processus de transcription pourrait être impliqué dans l’apparition des sites fragiles.
Pour la tester, les chercheurs ont utilisé la technique de cytométrie en flux. Cet outil leur a permis de trier les cellules selon leur avancement dans le cycle cellulaire (4) – cellules en phase G1 (transcription des gènes et croissance de la cellule), S (réplication de l’ADN), G2 (croissance et préparation à la division cellulaire) puis M (division cellulaire). Et, il est apparu que la transcription des très grands gènes dépasse largement la durée du cycle cellulaire, pour se terminer au début du cycle suivant, en phase G1 ou bien S. Un premier résultat étonnant : jusqu’à présent, il était admis, chez les mammifères, que la transcription des gènes avait lieu durant un même cycle cellulaire, et majoritairement en phase G1.
Comme la réplication survient pendant la phase S, les chercheurs ont soupçonné une interférence entre transcription et réplication pour expliquer les cassures sur les très grands gènes des mammifères. Ils ont donc étudié le processus de réplication sur ces gènes. Résultat : la réplication dans la région des sites fragiles survient à la fin de la phase S, alors que la transcription est encore en cours dans ces mêmes régions ! Cette découverte bouscule les connaissances actuelles en génétique. En effet, avant ces travaux, il était généralement admis que les machineries de transcription et de réplication de l’ADN ne pouvaient pas se rencontrer chez les mammifères.
Pour aller plus loin, l’équipe a cherché ensuite à savoir précisément ce qui pouvait fragiliser l’ADN quand réplication et transcription sont concomitantes. Ils ont mis en évidence des structures en boucle qui perdurent, dues à l’hybridation de l’ADN avec la molécule d’ARN produite lors de la transcription. Ce sont ces boucles ADN/ARN qui déstabiliseraient l’ADN jusqu’à provoquer des cassures en cas de stress.
Primordiale, cette découverte ouvre de nouvelles perspectives de recherche en médecine : les fameuses boucles apparaissent comme de possibles cibles pour réduire l’instabilité génomique et l’apparition de tumeurs.
Notes
(1) Processus lors duquel l’ADN est copié en ARN. Cet ARN est ensuite « traduit » en protéines afin de faire fonctionner la cellule.
(2) Processus permettant à l’ADN de se dédoubler avant la division de la cellule.
(3) La taille de l’ADN se mesure en Kilobase (1 kb = 1000 bases d’ADN)
(4) Ensemble des quatre phases par lesquelles une cellule passe pour se diviser, constitué de G1 (phase de transcription des gènes et de croissance de la cellule), S (réplication de l’ADN), G2 (croissance et préparation à la division de la cellule), et M (division).