Une équipe de l’Institut de génétique et de biologie moléculaire et cellulaire (IGBMC – CNRS/Université de Strasbourg/Inserm) vient de mettre en évidence, à l’échelle atomique, la structure tridimensionnelle du ribosome humain complet et les interactions fines qui y ont lieu. Ces résultats, obtenus grâce à une technologie unique en France, ouvrent la voie à de nouvelles explorations sur certains effets secondaires des antibiotiques et, à terme, pour le traitement de maladies liées aux dysfonctionnements du ribosome et à la dérégulation de la synthèse des protéines. Ces travaux sont publiés dans Nature le 22 avril 2015.
Les ribosomes sont de grands complexes constitués de protéines et d’ARN repliés ensemble qui, au sein des cellules de tous les êtres vivants, interviennent comme des nano-machineries moléculaires dans l’expression des gènes et la bio-synthèse des protéines. La structure des ribosomes de différentes espèces était déjà précisément connue à l’échelle atomique, mais déterminer celle particulièrement complexe du ribosome humain restait un défi majeur à relever.
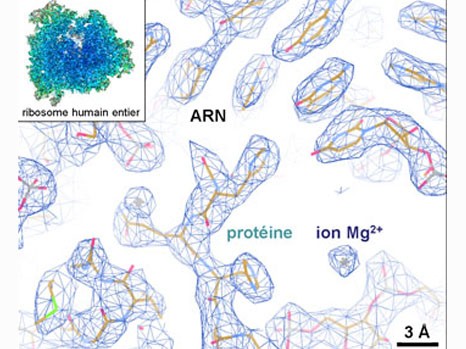
L’équipe de Bruno Klaholz, à l’IGBMC (CNRS/Université de Strasbourg/Inserm) vient de visualiser la structure atomique du ribosome humain complet avec une résolution supérieure à 3 angströms (0,3 nanomètres). Le modèle obtenu représente les 220 000 atomes qui constituent les deux sous-unités du ribosome et permet, pour la première fois, d’explorer son agencement en détail, de voir et d’identifier les différents acides aminés et nucléotides en 3 dimensions. Les chercheurs se sont plus particulièrement intéressés aux différents sites de liaison et aux interactions fines qui y ont lieu. Leurs travaux révèlent par exemple qu’après avoir livré les acides aminés qu’ils transportaient, les ARN de transfert continuent à interagir avec le ribosome dans un site particulier (le site de sortie des ARNt). Ils mettent également en lumière la dynamique des deux sous-unités du ribosome qui tournent légèrement sur elles-mêmes au cours du processus de bio-synthèse des protéines, entrainant un fort remodelage de la configuration 3D de la structure à leur interface.
Ces résultats ont été rendus possibles par un ensemble de technologies de pointe. Les échantillons, hautement purifiés puis congelés, ont été visualisés par cryo-microscopie électronique. Cette méthode permet de travailler sur des objets figés dont l’orientation ne change pas et dont la structure et les fonctions biologiques sont préservées. Une combinaison de traitement d’images et de reconstruction 3D appliquée aux images obtenues par le cryo-microscope électronique nouvelle génération de l’IGBMC – unique en France – a abouti à ce degré de précision rare.
Cette connaissance fine de la structure et de la dynamique du ribosome humain complet ouvre la voie à de nouvelles explorations majeures. Il est désormais envisageable d’étudier les effets secondaires de certains antibiotiques, destinés à s’attaquer aux ribosomes bactériens, qui peuvent cibler « par erreur » le ribosome humain. La constitution d’un répertoire des sites de liaison existants est une première étape pour augmenter la spécificité des molécules thérapeutiques et éviter qu’elles ne se fixent au mauvais endroit. A terme, ces résultats pourront également être utilisés pour la mise au point de traitement de maladies liées aux dysfonctionnements du ribosome et à la dérégulation de la synthèse des protéines. Dans le cas des cancers par exemple, pouvoir cibler les ribosomes des cellules malades permettrait de réduire leurs taux de synthèse de protéines. Exemple d’éléments tridimensionnels qui ont pu être distingués au sein de la structure atomique du ribosome humain complet (en encadré). La résolution (de l’ordre de l’angström) permet de déterminer s’il y a interaction ou non entre les différents éléments. © H. Khatter, A.G. Myasnikov, S. K. Natchiar & B.P. Klaholz