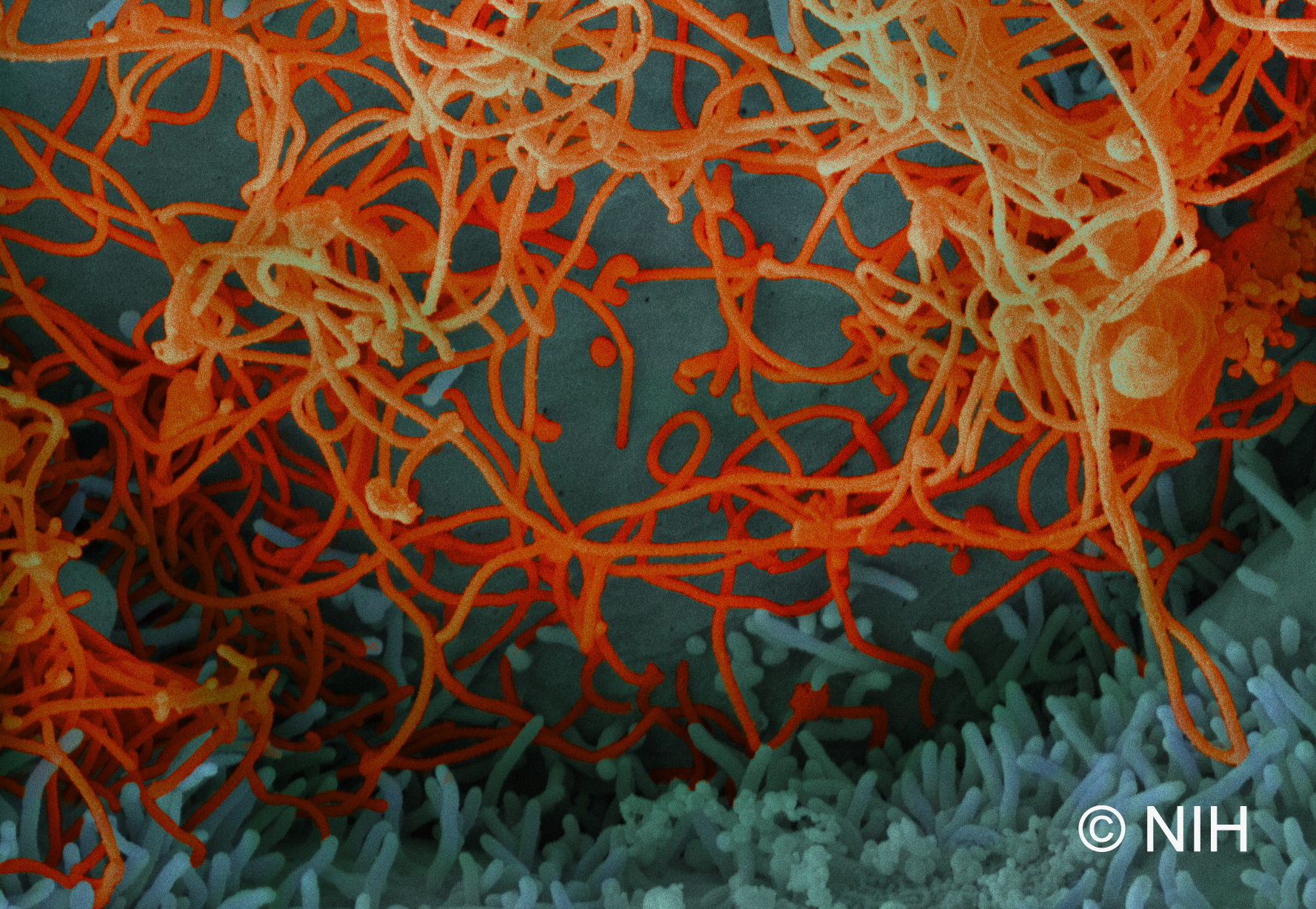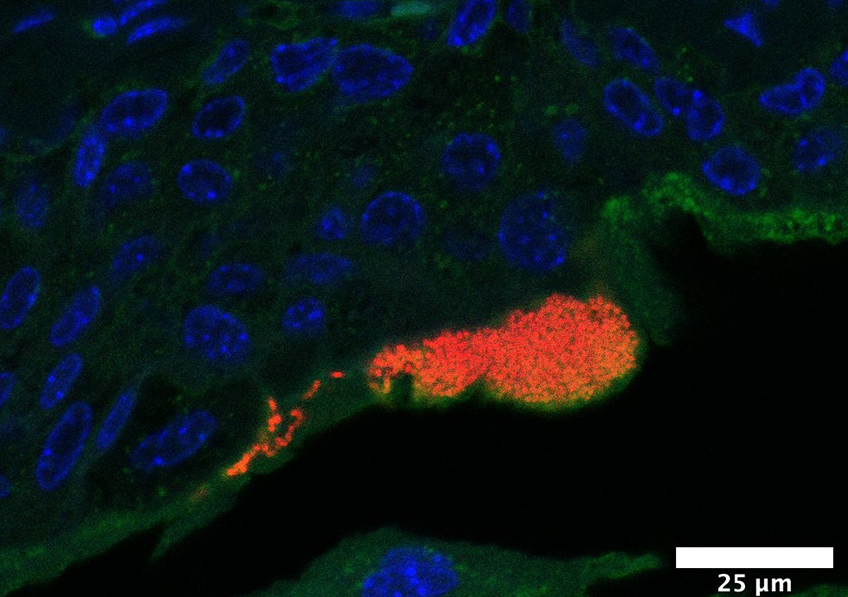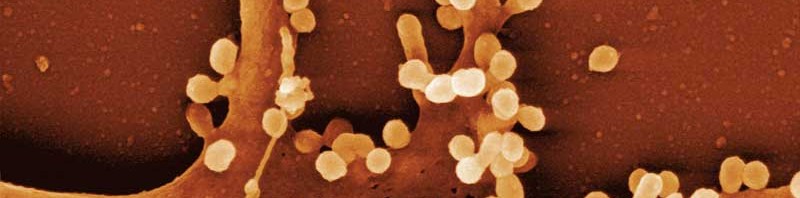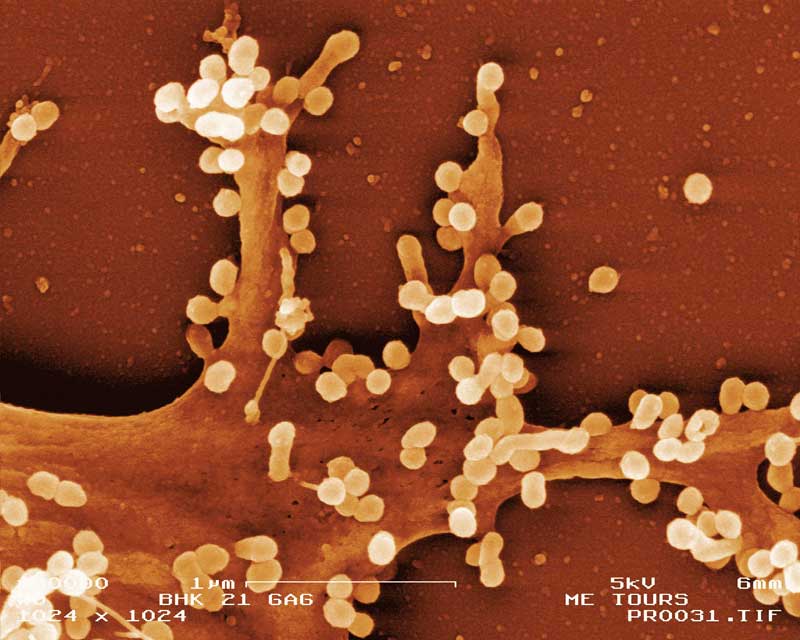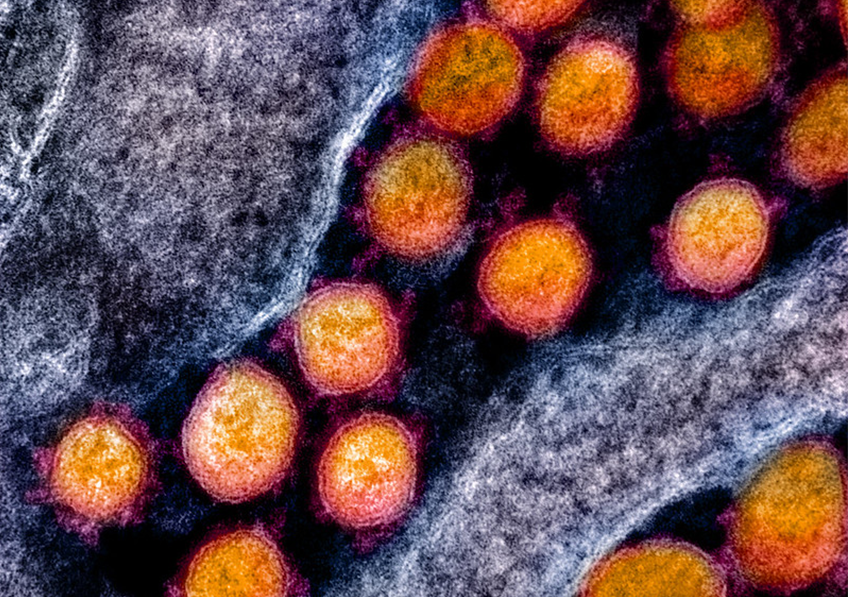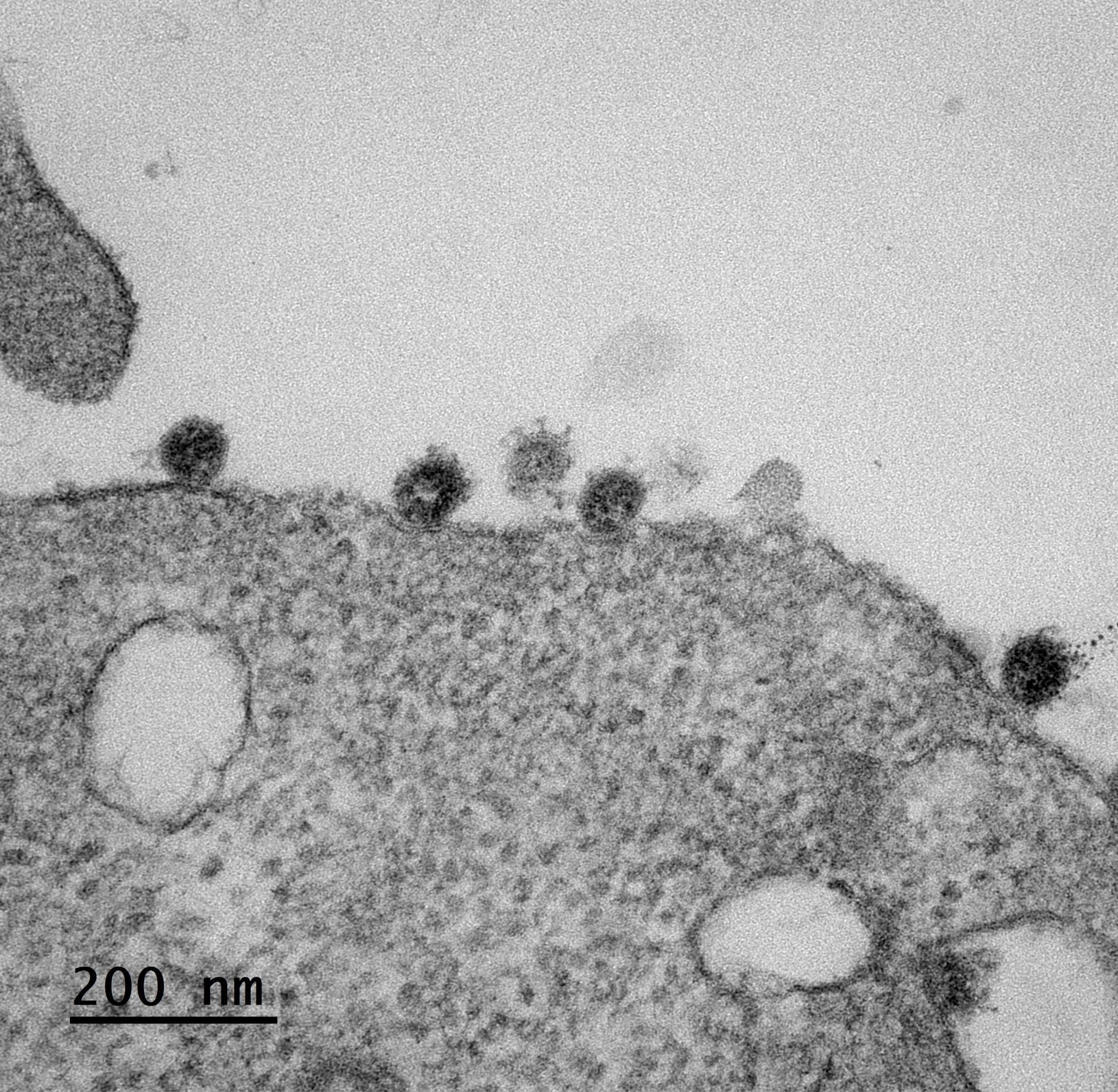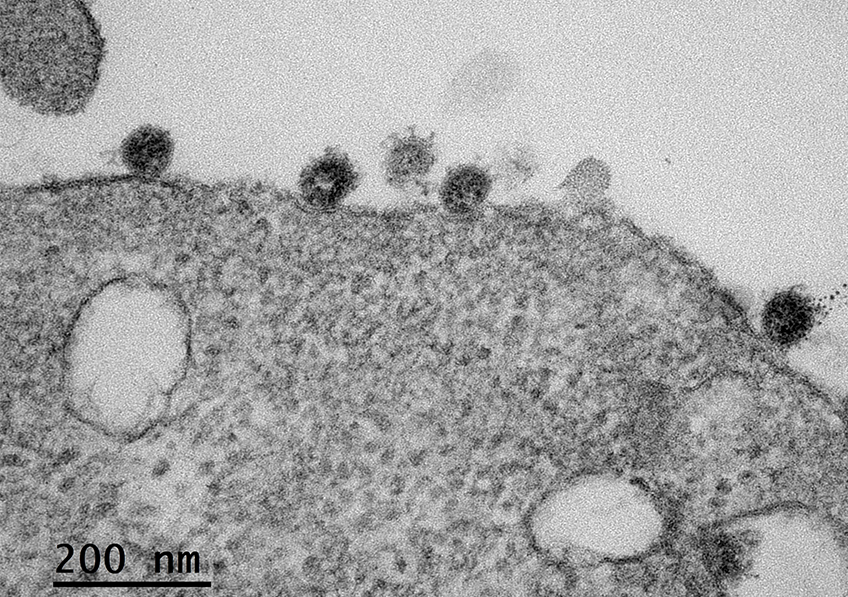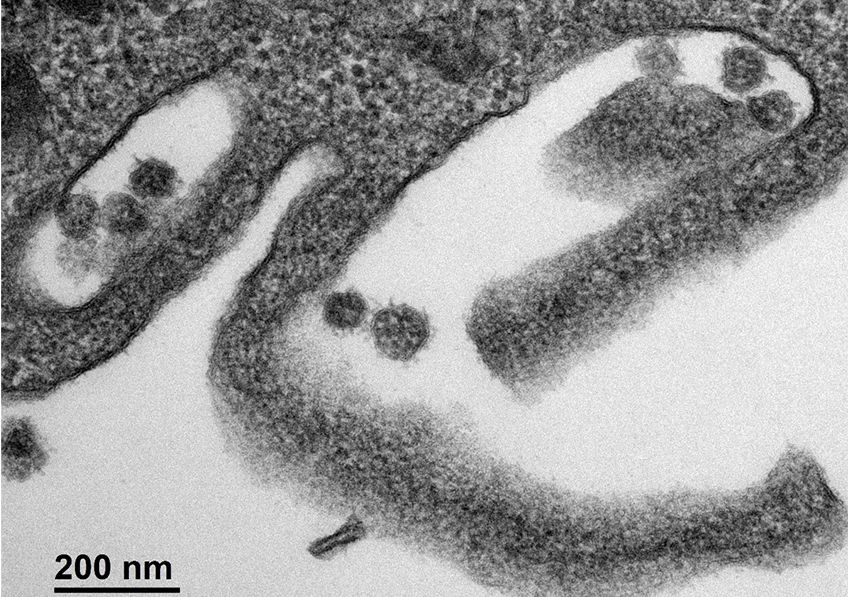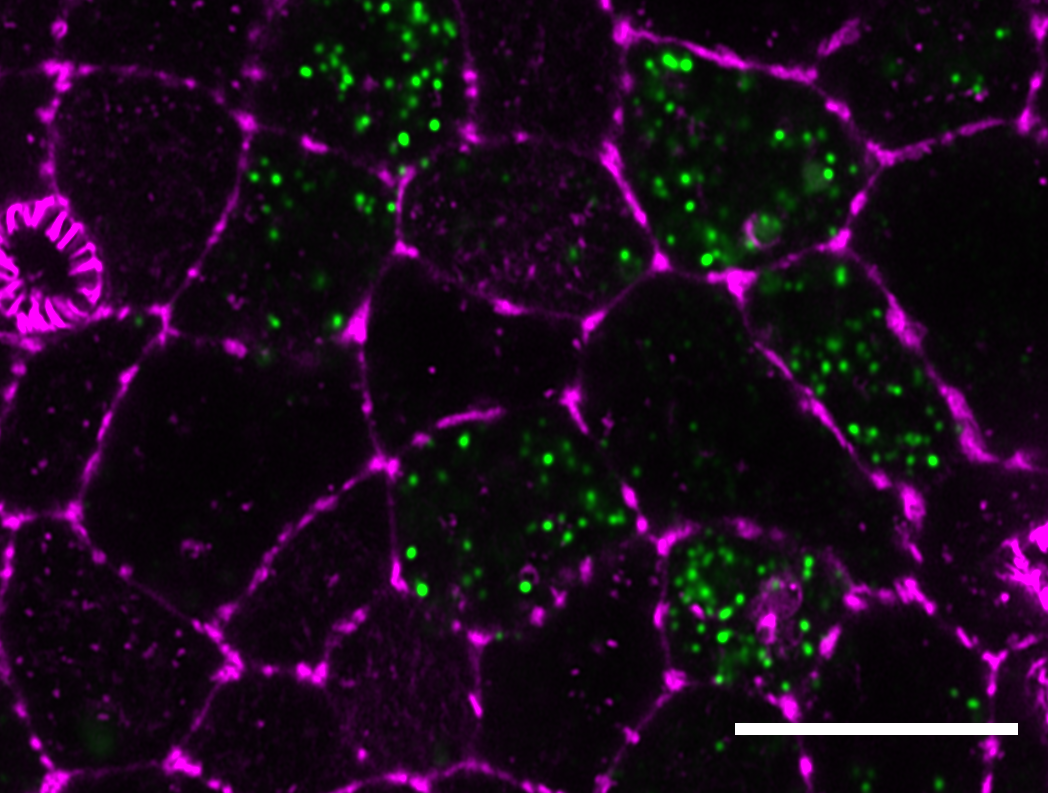
Cellules infectées par le SARS-CoV-2 (marqué en vert). L’échelle de taille correspond à 10µm. © Joe McKellar
Les cellules cibles du SARS-CoV-2 dans les voies respiratoires produisent des molécules antivirales suite à l’infection, mais trop tardivement pour empêcher la réplication du virus. C’est le constat que dressent les chercheurs et chercheuses de l’Inserm, du CNRS et de l’Université de Montpellier à l’Institut de recherche en Infectiologie de Montpellier. Leurs résultats sont publiés dans la revue Journal of Virology.
Avec son équipe, la chercheuse Inserm Caroline Goujon travaille de longue date sur les virus à l’Institut de Recherche en infectiologie de Montpellier (CNRS/université de Montpellier). Au début de l’épidémie de Covid-19, elle et son collègue Olivier Moncorgé décident de réorienter leurs recherches sur le nouveau coronavirus SARS-CoV-2 et lancent plusieurs projets dont l’un fondamental, destiné à comprendre la réponse des cellules cibles au moment de l’infection.
Alors que la communauté scientifique continue encore à se poser des questions sur le SARS-CoV-2, bâtir des connaissances sur les phases précoces de l’infection pour comprendre comment le virus pénètre dans l’organisme et comment il peut être bloqué est en effet essentiel.
A la recherche des interférons
Dans leur récente étude, les scientifiques décrivent cette réponse des cellules cibles dans l’épithélium respiratoire. Ils ont d’abord mis des cellules d’épithéliums respiratoires au contact du virus et analysé la multiplication de ce dernier dans les heures qui ont suivies. En parallèle, ils ont recherché la présence d’interférons, des molécules antivirales naturellement produites par les cellules en cas d’infection.
Ils ont constaté une augmentation rapide de la charge virale après 48 heures et une production importante de deux types d’interférons (les interférons de type I et de type III) entre 48 et 72 heures après l’infection. La production de ces molécules est classiquement déclenchée par certaines protéines des cellules hôtes – dites protéines sentinelles – car chargées de détecter la présence des virus.
Les chercheurs ont ensuite supprimé les gènes codants les principales protéines sentinelles avec la technique CRISPR-Cas9. Ils ont constaté que l’absence de l’un de ces gènes, MDA-5, empêchait la production des interférons. Cependant, ce phénomène n’avait pas d’impact sur la réplication virale. « Avec ou sans la production de ces interférons, qui ont pourtant pour but de contrecarrer le virus, la réplication virale avait lieu de la même manière dans notre modèle de cellules d’épithélium », clarifie Caroline Goujon.
Pourtant, les travaux de l’équipe et de nombreux autres groupes montrent que la mise en contact des cellules cibles avec ces mêmes interférons dans les heures précédant l’infection réduit énormément la capacité du virus à se répliquer : le taux de réplication est divisé par dix au minimum.
« L’activité antivirale des interférons n’est donc pas à remettre en cause contre le SARS-CoV-2. Leur inefficacité dans notre modèle est due à un problème de « timing ». Leur libération survient trop tard pour bloquer la réplication virale », explique-t-elle. Pour avoir un rôle protecteur et éviter la réplication virale, il semble important que leur production intervienne plus précocement.
Ouvrir des pistes de recherche thérapeutique
Ces travaux dessinent par ailleurs de nouvelles perspectives contre les formes sévères de Covid. « Nous savons grâce à des études précédentes que les niveaux d’interférons naturels sont bas chez les patients souffrant de forme grave de Covid-19 par rapport à ceux présentant une pathologie moins sévère. Stimuler précocement la production d’interférons par l’organisme en activant la voie MDA-5 pourrait permettre de limiter le risque de développement de formes sévères chez certains patients », ajoute la chercheuse.
Certaines équipes de recherche ont d’ores et déjà commencé à étudier l’administration précoce d’interférons dans le cadre de plusieurs essais cliniques. Caroline Goujon et ses collègues ont de leur côté entamé la suite du projet : identifier les gènes des cellules cibles dont l’expression est stimulée par l’infection et qui contribuent à freiner la réplication virale avec peut-être à la clé de nouvelles pistes thérapeutiques.