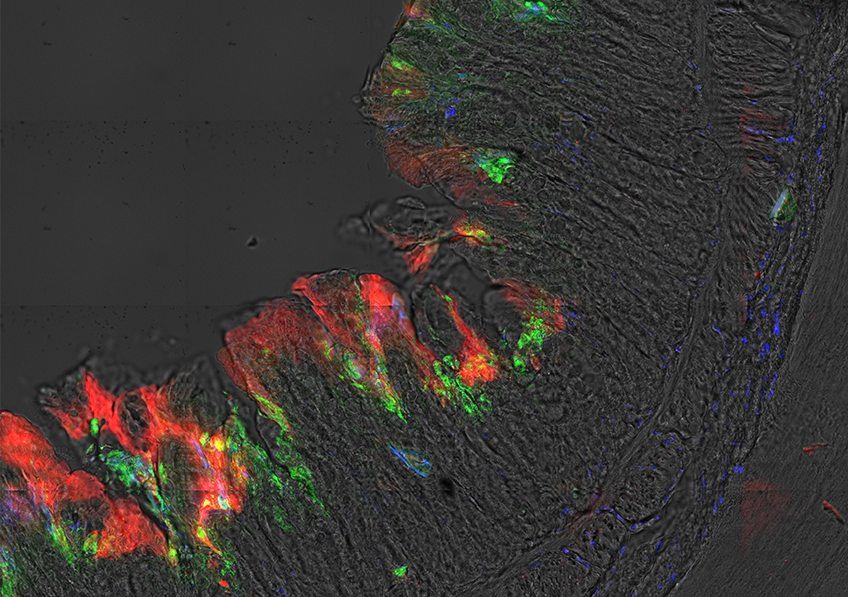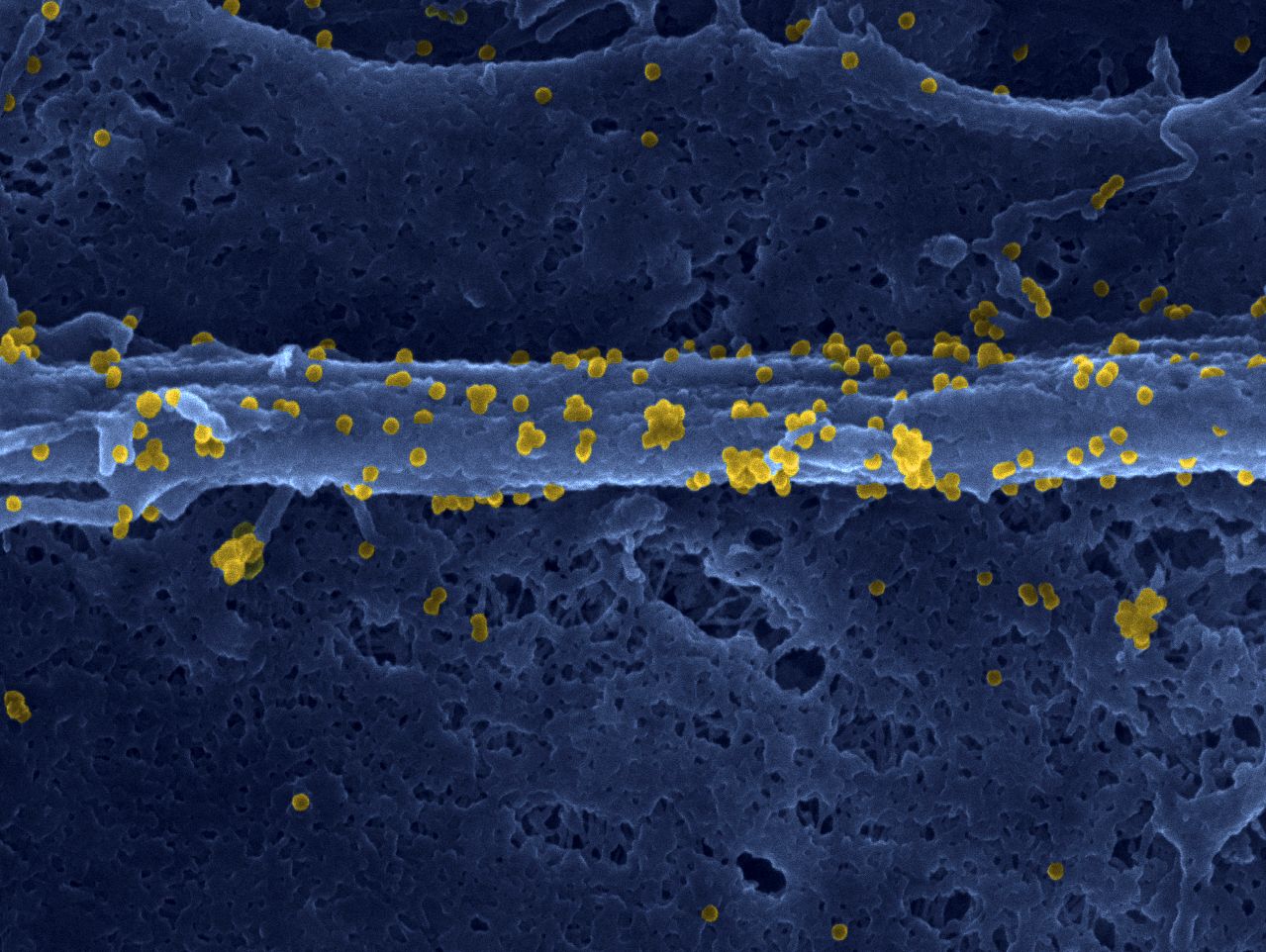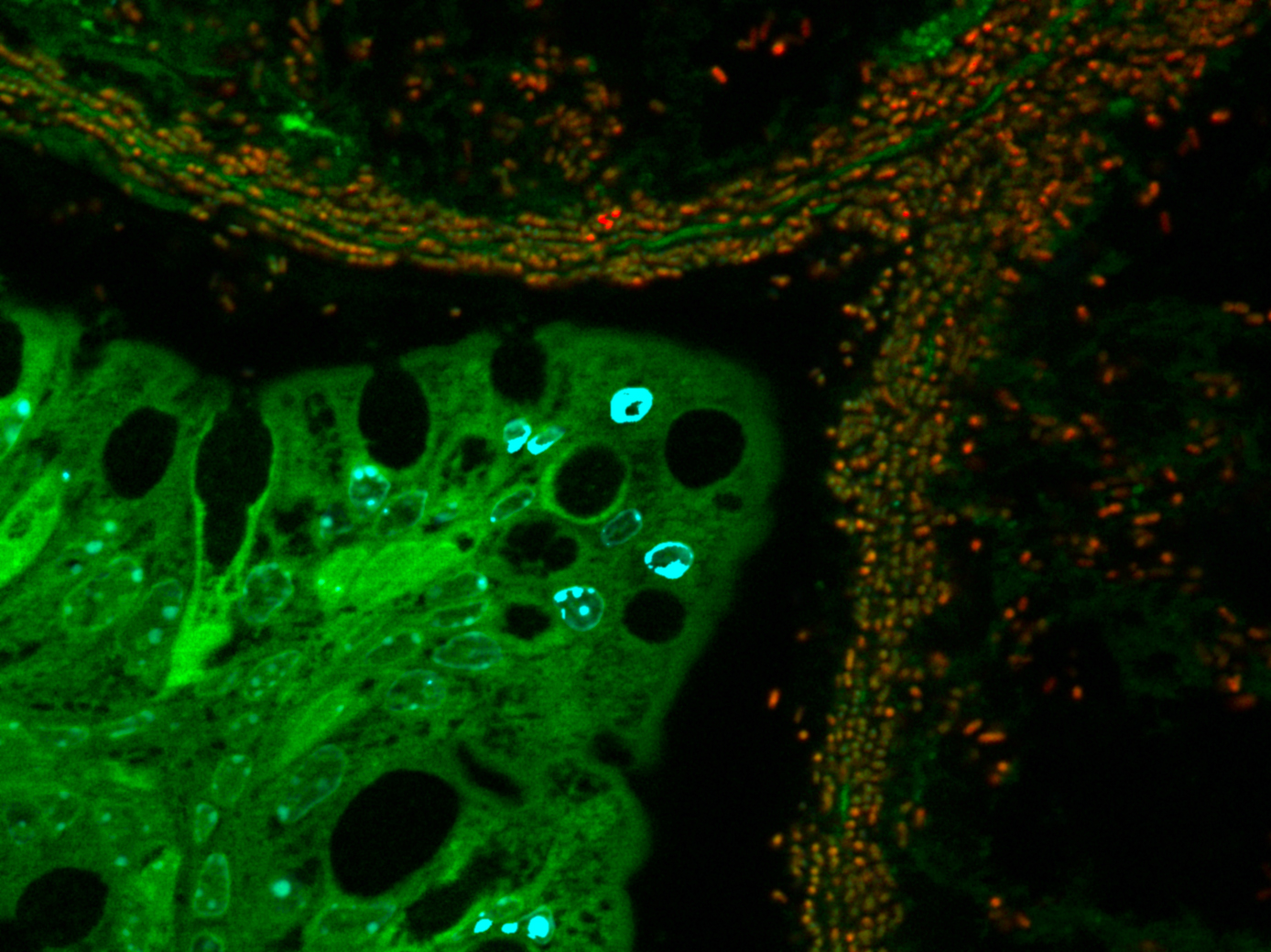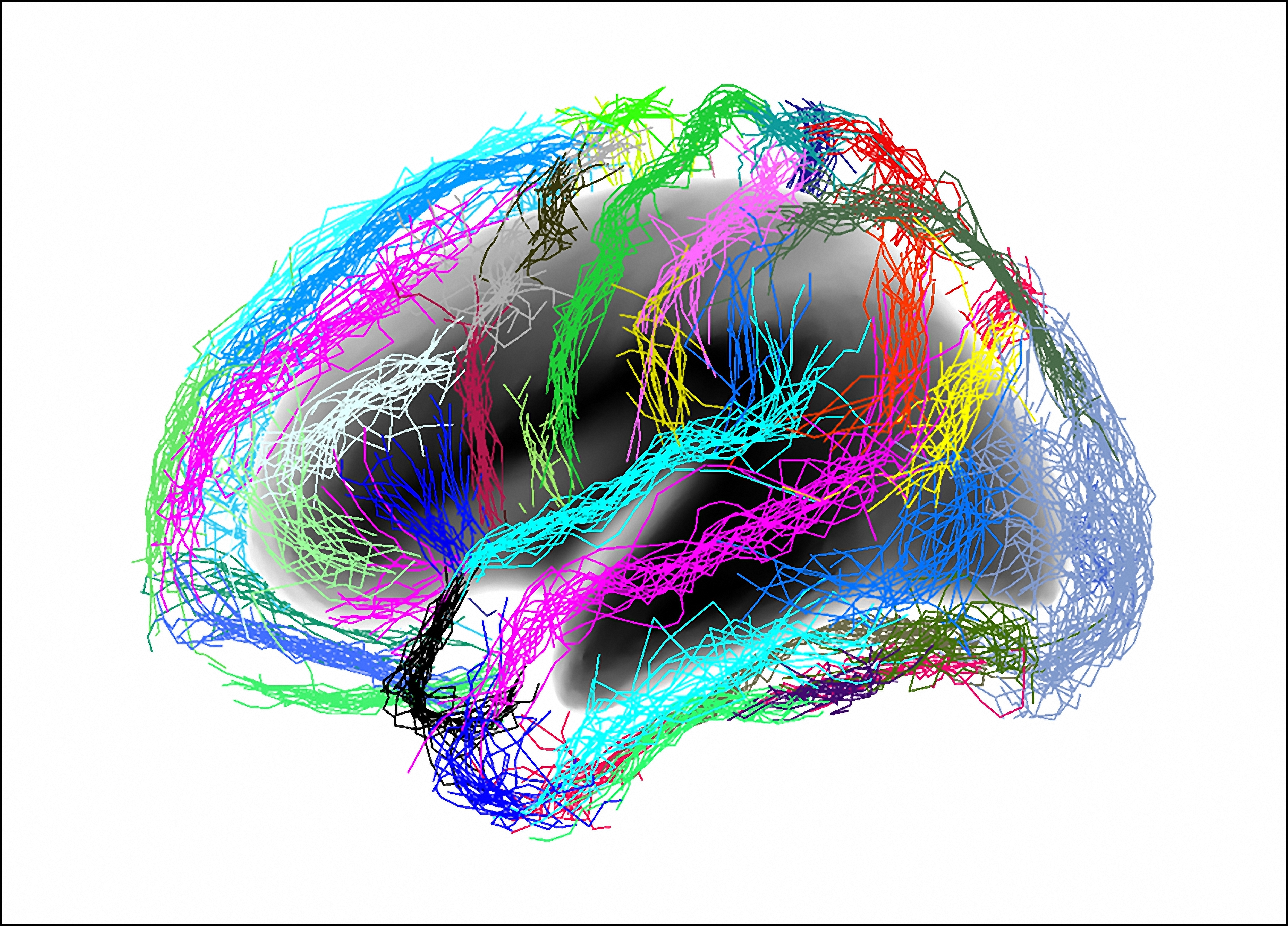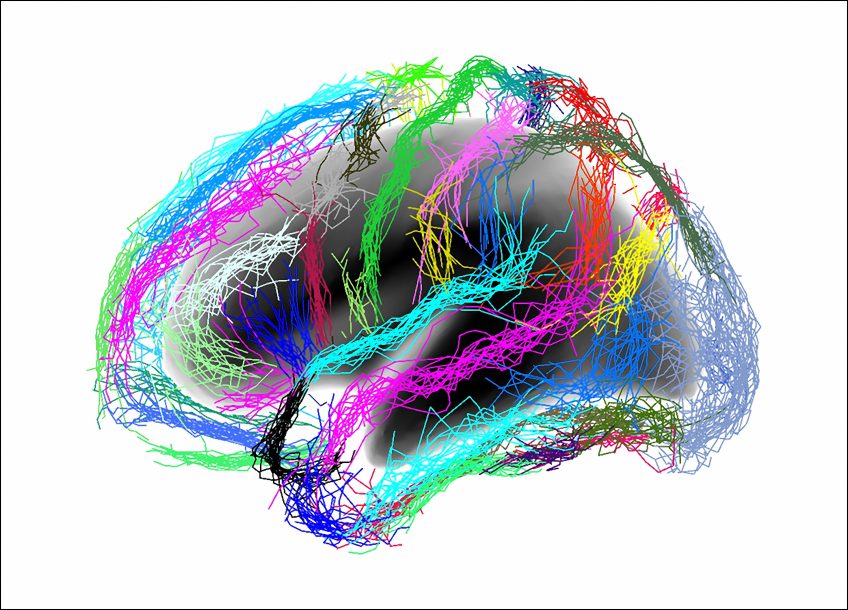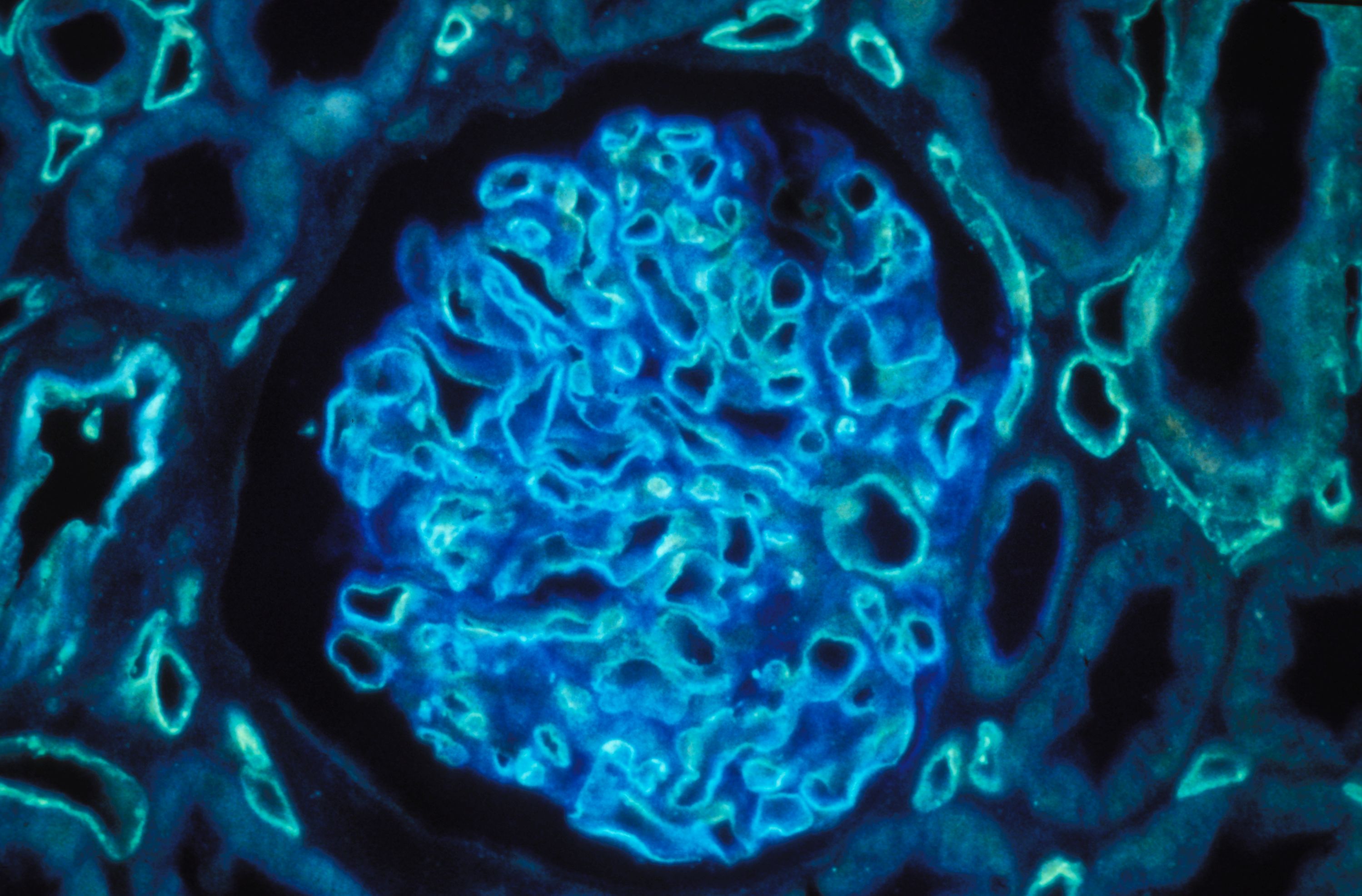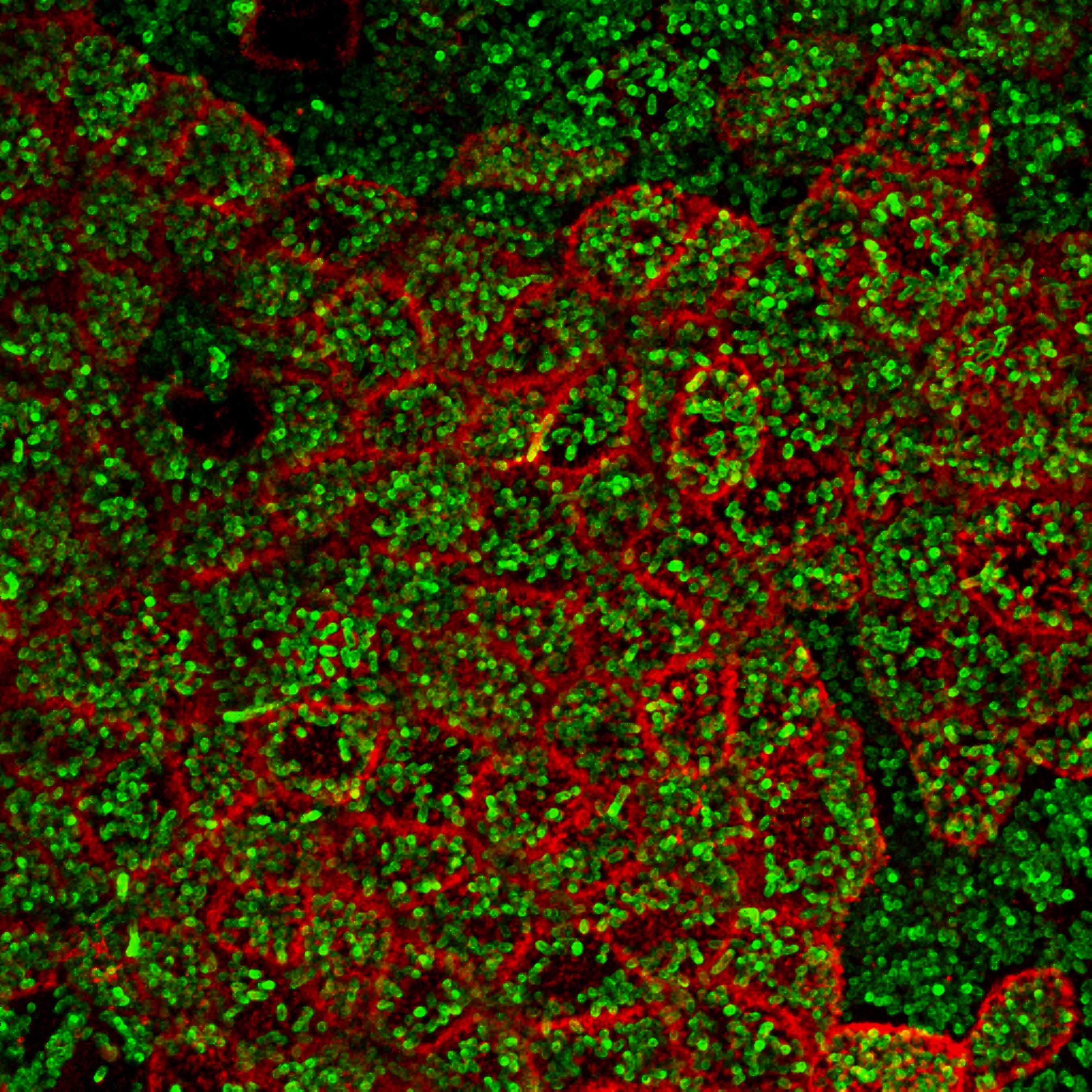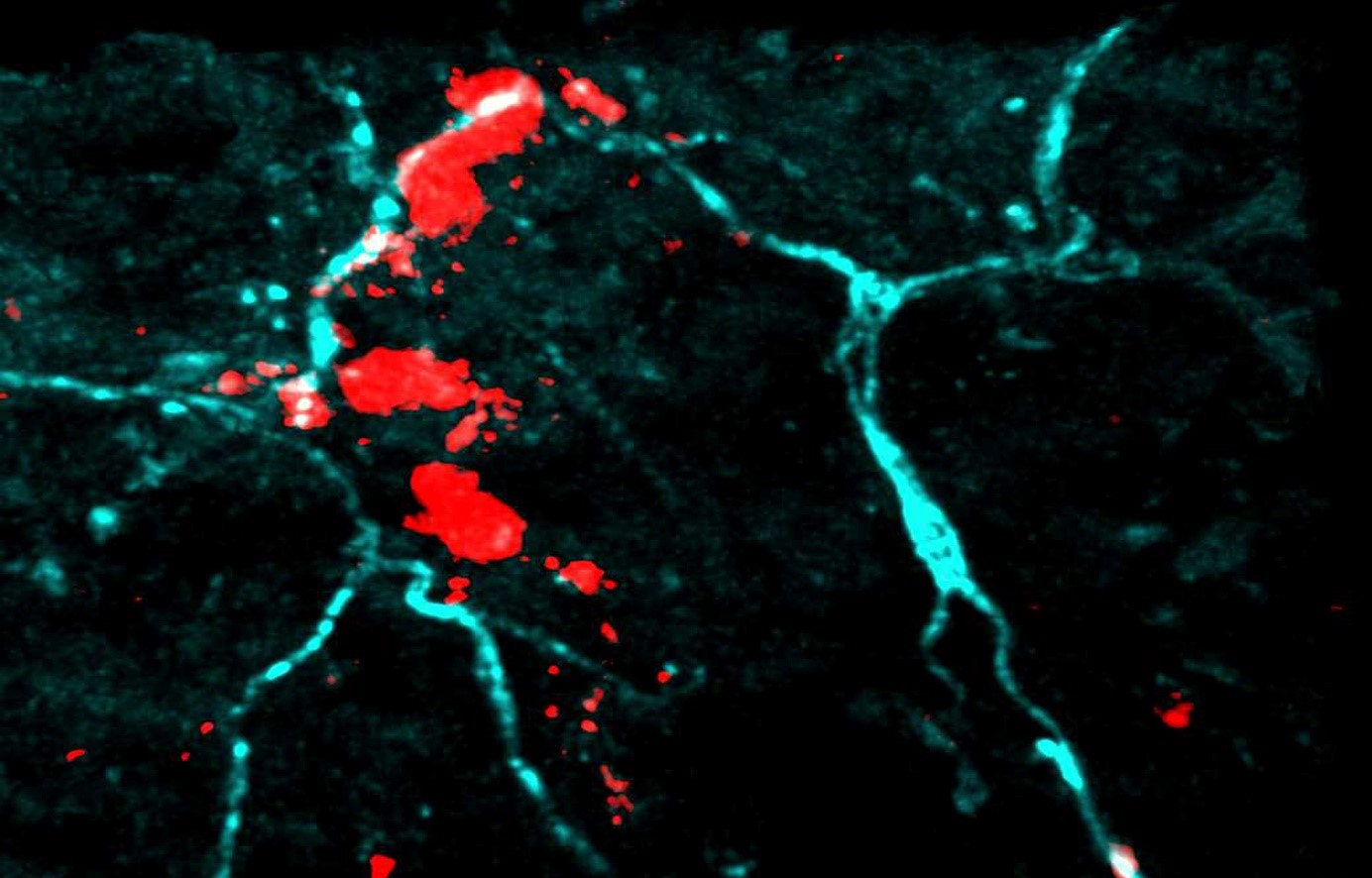
Les mastocytes et les neurones sensitifs s’agglutinent en « grappe » sous la peau. ©Nicolas Gaudenzio
La dermatite atopique, ou eczéma atopique, touche principalement les nourrissons et les enfants qui présentent alors une hypersensibilité aux allergènes de l’environnement. Maladie cutanée évoluant par poussées, elle est souvent traitée avec des anti-inflammatoires locaux. Une nouvelle étude dirigée par le chercheur Inserm Nicolas Gaudenzio, de l’Unité différenciation épithéliale et autoimmunité (UDEAR – Inserm / UT3 Paul Sabatier), en collaboration avec ses collègues de l’Université de Stanford (États-Unis) montre que les cellules immunitaires et les neurones sensitifs interagissent dans la peau pour former des unités capables de détecter les allergènes et de déclencher l’inflammation. Une découverte qui permet de mieux comprendre le fonctionnement de la dermatite atopique et d’envisager de nouvelles pistes thérapeutiques. Les résultats sont désormais publiés dans la revue Nature Immunology.
Peau sèche, douleur, démangeaisons… La dermatite atopique affecte le quotidien de près de 20 % des enfants, et jusqu’à 5 % des adultes. La qualité de vie de ces patients peut en être très dégradée.
Plusieurs travaux ont démontré que des facteurs génétiques sont impliqués dans le développement de cette maladie chronique inflammatoire de la peau, et qu’ils sont probablement à l’origine d’une altération de la barrière cutanée. Les allergènes présents dans l’environnement, des poussières au pollen en passant par les acariens, peuvent alors pénétrer dans le derme et stimuler le système immunitaire, qui réagit anormalement à cette « menace » en déclenchant l’eczéma.
Néanmoins, les mécanismes de l’hypersensibilité aux allergènes et de l’hyperactivité du système immunitaire chez les patients souffrant de dermatite atopique ne sont pas encore tous bien connus. Menée par le chercheur Inserm Nicolas Gaudenzio, la jeune équipe « IMMCEPTION » étudie la manière dont le système immunitaire interagit avec les neurones sensitifs pour réguler les processus inflammatoires dans la dermatite atopique.
Les chercheurs s’appuient notamment sur des données cliniques déjà disponibles qui soulignent que le sang des patients atteints de cette pathologie contient de nombreux neuropeptides, messagers chimiques porteurs des messages nerveux, et dont la quantité est corrélée avec la sévérité de la maladie. Que ces neuropeptides soient identifiés dans le sang indique que les neurones sensitifs sont activés. Par ailleurs, une quantité d’enzymes reflétant la présence de mastocytes est aussi retrouvée dans le sang de ces patients, les mastocytes étant des cellules immunitaires présentes dans la peau et essentielles à la modulation des processus inflammatoires et allergiques.
Partant de ces constats, Nicolas Gaudenzio et son équipe ont décidé de s’intéresser à l’interaction entre neurones sensitifs et mastocytes, et publient à présent leurs résultats dans le journal scientifique Nature Immunology.
Les scientifiques ont travaillé avec des modèles animaux de dermatite atopique. Sous la peau de souris présentant des signes de réactions inflammatoires, ils ont observé que les mastocytes et les neurones sensitifs s’agglutinaient, se regroupant en « unités neuro-immunes sensorielles », dont la forme n’est pas sans rappeler celle d’une grappe de raisin. « Les mastocytes et les neurones se collent les uns aux autres au niveau du derme. On ne sait pas encore quelles sont les interactions moléculaires qui les lient les uns aux autres, mais on a quantifié les distances entre eux, et elles sont minimes », souligne Nicolas Gaudenzio.
Les chercheurs ont ensuite montré que lorsque les souris sont exposées à des acariens, ces « unités neuro-immunes sensorielles » sont capables de détecter la présence de ces allergènes, ce qui déclenche une inflammation allergique.
A plus long terme, une telle découverte pourrait avoir des implications thérapeutiques concrètes. « Jusqu’à maintenant, les patients peuvent être soignés avec des traitements biologiques (biothérapies) mais ceux-ci traitent certainement la pathologie plus en aval, une fois que les poussées se sont déclarées. Nous pensons que nous avons mis le doigt sur un mécanisme déclencheur et nous voulons à présent poursuivre les recherches pour identifier de nouvelles molécules qui pourraient bloquer les interactions entre les mastocytes et les neurones sensitifs, et donc avoir une action thérapeutique bénéfique pour les patients », explique le chercheur.
Pour y parvenir, l’équipe devra d’abord caractériser plus précisément les interactions moléculaires au sein de ces unités, et analyser le rôle qu’elles jouent dans la modulation du système immunitaire.
« Une des questions à laquelle on va maintenant tenter de répondre, c’est à quoi servent ces unités mastocytes-neurones sensitifs. Elles doivent constituer un mécanisme de défense de l’organisme, étant donné qu’elles se retrouvent aussi chez les individus sains. Mais peut-être leur fonctionnement est-il altéré chez ceux qui souffrent de dermatite atopique et c’est ce qu’on va chercher à comprendre », conclut Nicolas Gaudenzio.