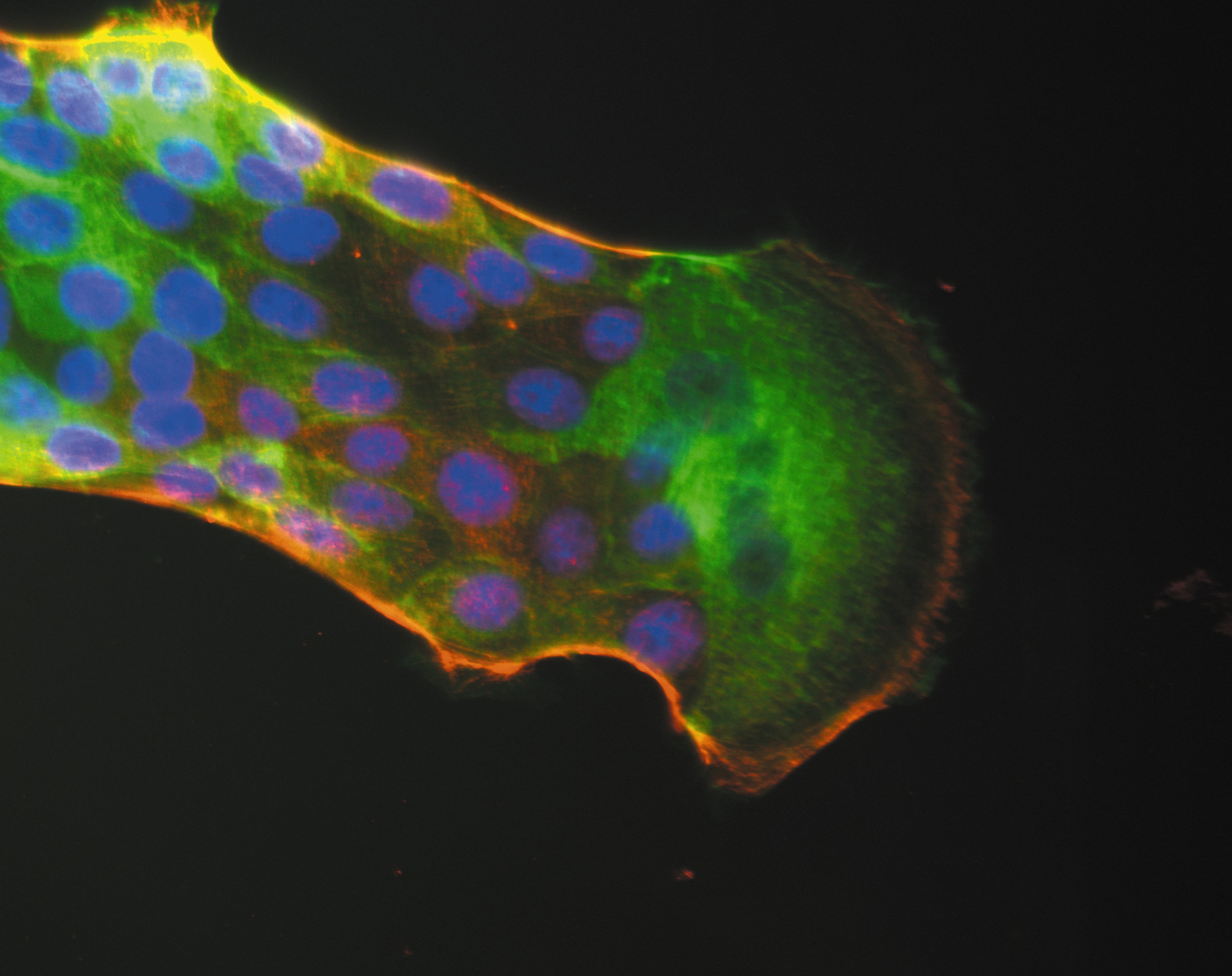
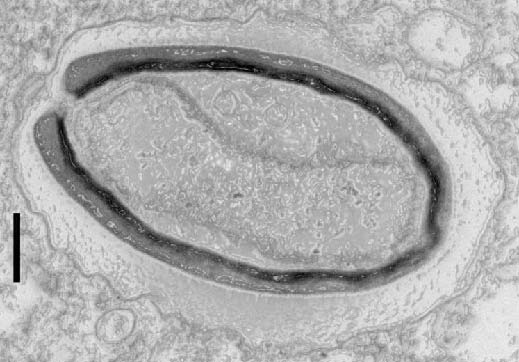
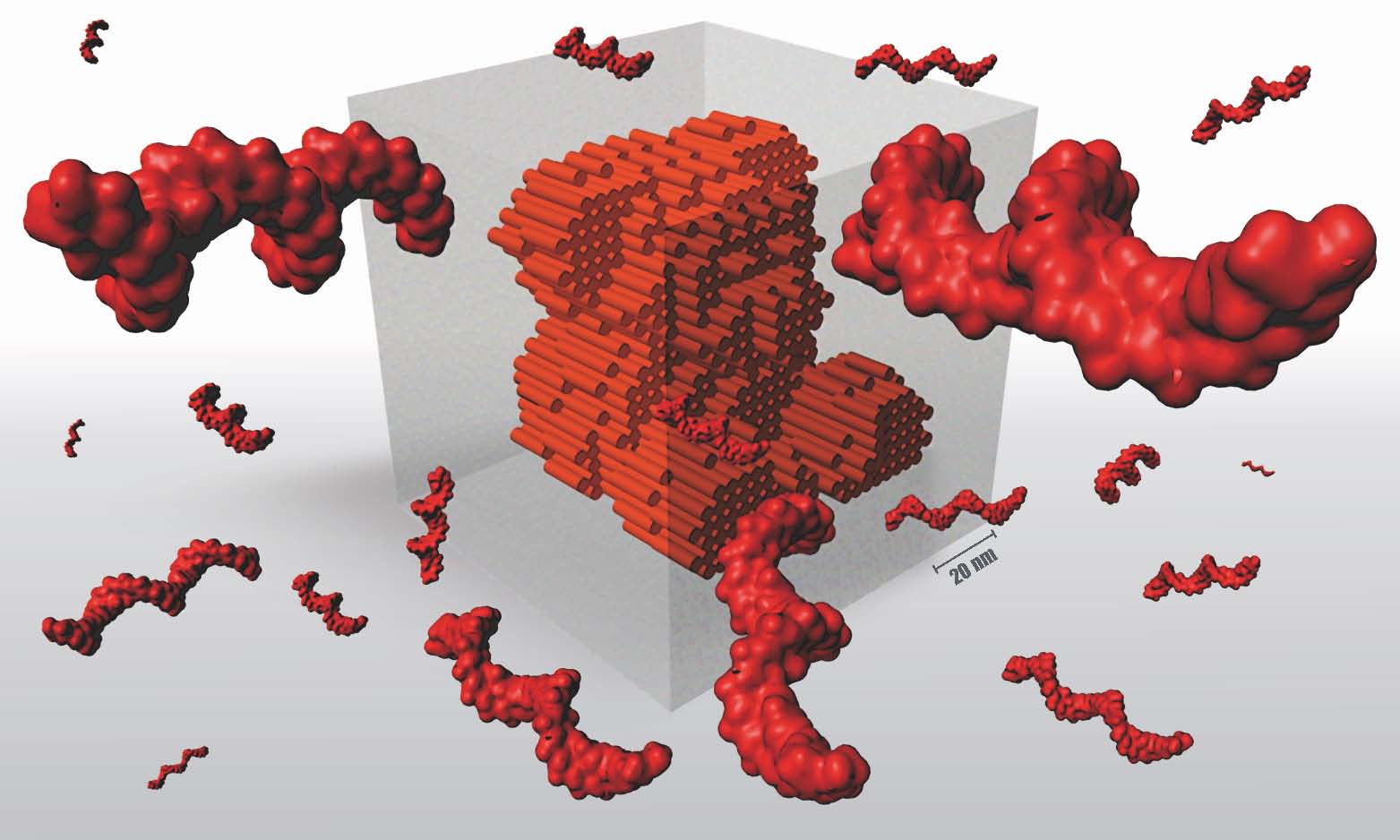
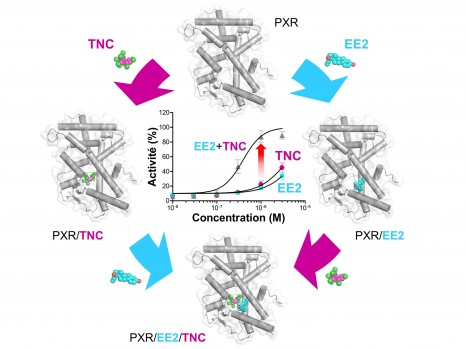
©Photo AdobeStock
Tout au long de l’évolution, le génome de la plupart des êtres vivants s’est complexifié grâce aux éléments transposables ou « gènes sauteurs », des fragments d’ADN capables de se déplacer ou de se copier d’un endroit à un autre sur les chromosomes. Des chercheurs de l’Inserm, du CNRS, d’Université Côte d’Azur et de l’Université de Montpellier ont pu capturer ces « gènes sauteurs » juste après leur déplacement et ont croisé leurs observations avec des bases de données déjà existantes. Leurs travaux, à paraître dans Molecular Cell, montrent que l’intégration des « gènes sauteurs » chez l’humain ne se ferait pas au hasard mais serait influencée par certaines propriétés du génome. Ces résultats ouvrent ainsi de nouvelles perspectives dans l’interprétation des données de séquençage de génome entier.
Les éléments transposables, aussi appelés « gènes sauteurs », sont de petits fragments d’ADN capables de se multiplier et de se déplacer dans les chromosomes de la plupart des organismes vivants. Cette prolifération a été tellement intense chez les mammifères et les primates, qu’ils constituent plus de la moitié de nos chromosomes ! Bien sûr, ils ne sautent pas tous en même temps, dans toutes nos cellules. Parmi toutes les copies présentes dans notre ADN, seule une petite fraction est toujours active. Toutes les autres sont des vestiges moléculaires qui reflètent des millions d’années d’évolution au cours desquelles les insertions néfastes ont été éliminées et celles bénéfiques conservées.
Chez l’humain, les gènes sauteurs les plus actifs sont les rétrotransposons L1. En sautant, ils peuvent altérer ou détruire des gènes et provoquer l’apparition de maladies génétiques comme des hémophilies ou des dystrophies musculaires. Les rétrotransposons L1 sont aussi particulièrement actifs dans certaines formes de cancers, et pourraient être impliqués dans le vieillissement cellulaire ou dans certaines maladies mentales.
Les rétrotransposons L1 ciblent-ils des régions chromosomiques spécifiques ou s’insèrent-ils au hasard ? Les équipes de Gaël Cristofari et Simona Saccani, directeurs de recherche à l’Inserm au sein de l’Institut de recherche sur le cancer et le vieillissement de Nice – Ircan (Inserm, CNRS, Université Côte d’Azur), et leurs collaborateurs à l’Université de Montpellier, sont parvenus, grâce à une technique de séquençage du génome dite « à haut débit », à capturer ces gènes sauteurs en pleine action juste après qu’ils aient sauté à une nouvelle position. En croisant leurs observations avec des banques de données génomiques et épigénomiques, les chercheurs ont identifié les caractéristiques du génome qui influencent l’intégration des rétrotransposons L1, la plus notable étant la réplication de l’ADN, ainsi que le rôle prépondérant des phénomènes de sélection naturelle après intégration.
« Jusqu’à présent, on savait que les rétrotransposons L1 ont tendance à s’accumuler dans certaines régions de nos chromosomes, notamment l’hétérochromatine. Mais on ne savait pas si cela reflète une attraction particulière pour ces régions, ou s’ils sont uniquement tolérés dans ces régions et éliminés ailleurs par sélection naturelle. Lorsqu’on sait où ils sautent et les copies qui sont conservées au cours de l’évolution, on peut découvrir – en négatif – les régions où ils peuvent faire des dommages », explique Gaël Cristofari.